GRUPOS DE INVESTIGACIÓN
Metaloproteínas
RESUMEN

- Vila, Alejandro Sede: CCT
Email: vila@ibr-conicet.gov.ar
- Hita, Francisco
LÍNEA DE INVESTIGACIÓN
Resistencia a antibióticos: Evolución, diversidad estructural y mecanismo catalítico de Metalo-β-Lactamasas
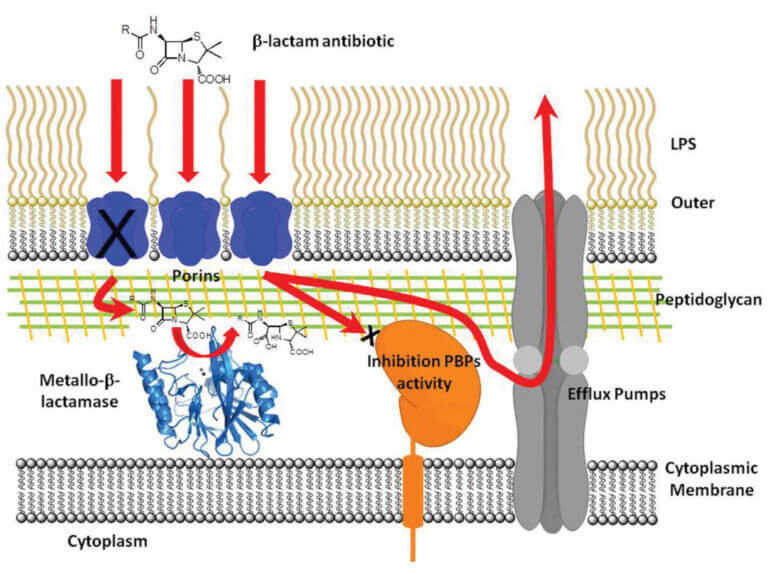
Los antibióticos β-lactámicos son los agentes quimioterapéuticos más empleados para el tratamiento de infecciones bacterianas. El principal mecanismo de resistencia a estos fármacos es la síntesis de β-lactamasas, enzimas que hidrolizan e inhiben la acción de estos antibióticos. Las metalo-β-lactamasas (MβLs) constituyen la generación más reciente de estas enzimas. Las MβLs poseen un amplio espectro de acción, ya que inactivan todos los antibióticos β-lactámicos, incluidos los carbapenemes (el último recurso empleado en la clínica).
La diseminación de genes que codifican estas enzimas en organismos oportunistas y patógenos es hoy de escala mundial. Este cuadro se agrava por la diversidad estructural de las distintas MβLs, que dificulta el diseño de un inhibidor eficiente para estas enzimas. La enzima NDM-1 (http://www.bbc.co.uk/news/health-10925411), en particular, está siendo diseminada en todo el mundo rápidamente, y no se cuenta con inhibidores de uso clínico para estas enzimas, lo que representa una amenaza para la salud mundial.
Nuestro objetivo es elucidar la relación estructura-función de estas enzimas mediante estudios bioquímicos, estructurales, mecanísticos y evolutivos, con el fin último de diseñar un inhibidor de aplicación clínica. Hasta el momento, hemos logrado: (1) proponer un mecanismo catalítico común para las MβLs a partir de la caracterización de un intermediario de reacción, (2) identificar la especie funcional in vivo de las mismas y (3) explorar sus posibles mecanismos evolutivos. En la actualidad, utilizamos estos conocimientos previos para diseñar y generar inhibidores de MβLs, bajo la hipótesis de que, a pesar de su diversidad estructural, las MβLs actúan utilizando el mismo mecanismo catalítico.
Nuestro grupo lleva adelante un estudio interdisciplinario, usando técnicas de biología molecular, bioquímica, biología estructural y enzimología moderna. Los estudios mecanísticos se realizan usando técnicas de mezcla rápida y varias espectroscopías para seguir cambios en el sitio activo durante el recambio catalítico en la escala de tiempo de los milisegundos, con el propósito de identificar intermediarios de reacción.
Utilizamos cristalografía y/o Resonancia Magnética Nuclear (RMN) para caracterizar el modo de unión de posibles inhibidores a estas enzimas. También usamos estrategias de evolución dirigida in vitro como un medio para pronosticar la evolución de estas enzimas en ambientes hospitalarios, y para entender de qué modo se fijan las mutaciones en el proceso evolutivo.
Director de Grupo: Vila, Alejandro
Cobre en sistemas biológicos: Control de la reactividad y mecanismo de inserción de sitios de cobre de transferencia electrónica
La respiración celular depende del control fino de una serie de procesos de oxido-reducción, que culminan en la reducción de oxígeno llevada a cabo por las citocromo c oxidasas. El cobre es un metal de transición esencial para la transferencia electrónica en estas enzimas, siendo a la vez tóxico a altas concentraciones. Por lo tanto, su concentración intracelular y mecanismos de transporte y almacenamiento deben estar finamente regulados.
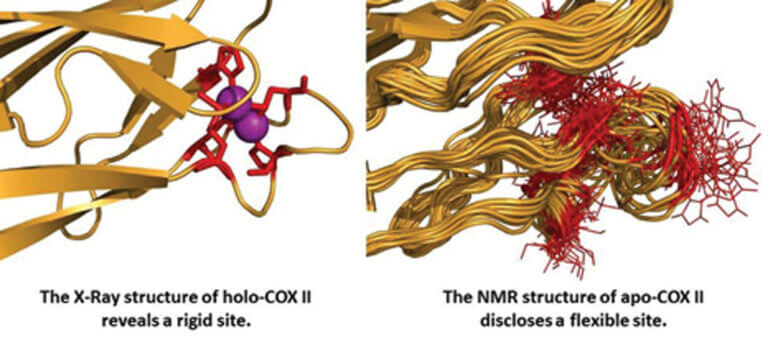
Nuestro grupo estudia: (1) los mecanismos biológicos de transporte e inserción de iones cobre en oxidasas terminales y (2) la regulación de la función de estos sitios mediante el ajuste fino de su estructura electrónica y tridimensional. Hemos identificado las proteínas involucradas en el transporte e inserción de cobre en el sitio CuA de citocromo oxidasas bacterianas, y caracterizado estructuralmente el mecanismo de unión a metal empleando RMN. Para estudiar la estructura electrónica de estos sitios, los métodos de biología estructural clásicos resultan insuficientes, ya que sólo proveen la estructura tridimensional.
En nuestro laboratorio utilizamos varias técnicas espectroscópicas (principalmente RMN) para analizar la distribución electrónica en sitios de cobre, y estudiar de qué modo la misma determina la transferencia de electrones a distancia. Hemos empleado esta estrategia para describir el ajuste fino de la reactividad en proteínas azules de cobre, y en la actualidad estamos aplicando nuestra experiencia en el estudio del sitio de cobre CuA en citocromo c oxidasas. Hemos propuesto que la alta eficiencia del sitio CuA en transferencia electrónica a distancia se debe fundamentalmente a la presencia de estados electrónicos excitados de baja energía que se encuentran poblados a temperatura ambiente.
IMÁGENES DE NUESTRAS INVESTIGACIONES
PUBLICACIONES Y PATENTES
M.N. Lisa, A.R. Palacios, M. Aitha, M. M. González, D.M.Moreno, M.W. Crowder, J. Spencer, D.L. Tierney, L.I. Llarrull and A. J. Vila
M.N. Lisa, A.R. Palacios, M. Aitha, M. M. González, D.M.Moreno, M.W. Crowder, J. Spencer, D.L. Tierney, L.I. Llarrull and A. J. Vila
P.Hinchliffe, M.M. González, M. F. Mojica, J. M. González, V. Castillo, C. Saiz, M. Kosmopoulou, C. L. Tooke, L. I. Llarrull, G. Mahler , R. A. Bonomo , A.J. Vila*, J. Spencer*
P.Hinchliffe, M.M. González, M. F. Mojica, J. M. González, V. Castillo, C. Saiz, M. Kosmopoulou, C. L. Tooke, L. I. Llarrull, G. Mahler , R. A. Bonomo , A.J. Vila*, J. Spencer*
L.J. González, G. Bahr, T.G. Nakashige, E.M. Nolan, R.A. Bonomo and A.J.Vila
L.J. González, G. Bahr, T.G. Nakashige, E.M. Nolan, R.A. Bonomo and A.J.Vila
M.N. Morgada, L.A. Abriata, C. Cefaro, K.Gajda, L. Banci and A.J.Vila
M.N. Morgada, L.A. Abriata, C. Cefaro, K.Gajda, L. Banci and A.J.Vila
M.N.Morgada, L.A.Abriata, U.Zitare, D.Álvarez-Paggi, D.H.Murgida and A. J.Vila
M.N.Morgada, L.A.Abriata, U.Zitare, D.Álvarez-Paggi, D.H.Murgida and A. J.Vila
Sede CCT Rosario
Ocampo y Esmeralda, Predio CONICET-Rosario
2000 Rosario, Santa Fe, Argentina
Tel. 54-341-4237070 / 4237500 / 4237200
Sede Facultad de Ciencias Bioquímicas y Farmacéuticas
Universidad Nacional de Rosario - Suipacha 531
2000 Rosario, Santa Fe, Argentina
Tel. +54 341 4350596 / 4350661 / 4351235
🔬 El IBR suma 9 proyectos seleccionados en Investigación Orientada 2025 de @ProduccionSF y @CienciaSantaFe.
Biotecnología, salud y sostenibilidad para fortalecer el vínculo entre ciencia, innovación y desarrollo territorial.

