GRUPOS DE INVESTIGACIÓN
Biofísica del Reconocimiento Molecular
RESUMEN

- Rasia, Roldolfo Sede: CCT
Email: rasia@ibr-conicet.gov.ar
- Cenizo, Zoe
LÍNEA DE INVESTIGACIÓN
Plegamiento de proteínas inducido en la regulación génica de plantas
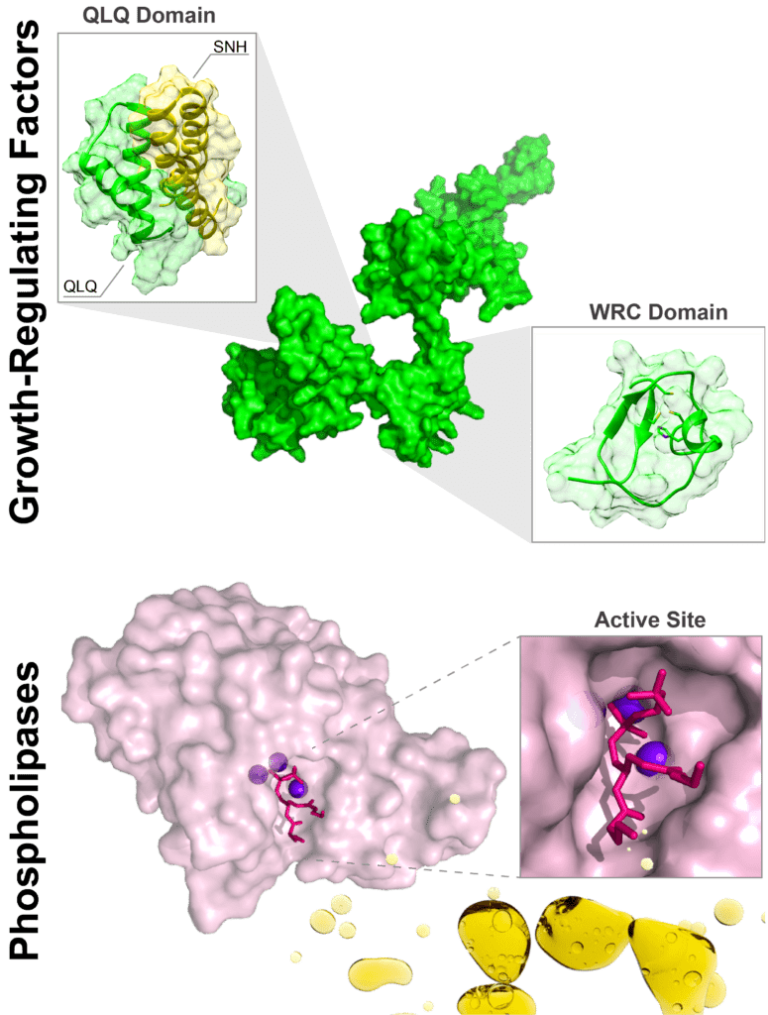
La expresión génica en organismos eucariotas está finamente regulada a través de la acción de proteínas específicas: los factores de transcripción, que unen secuencias de ADN y controlan de esta manera la transcripción de los genes adyacentes que son a su vez auxiliados por coactivadores transcripcionales, que contribuyen a la reorganización de la cromatina. En este proyecto estudiamos la función del sistema GRF:GIF de plantas. En Arabidopsis thaliana la familia GRF tiene 9 miembros y la famila GIF tiene 3. Las proteínas en estas dos familias son mayormente desordenadas. Los factores de transcripción GRF reconocen el ADN a través de sus dominios WRC. A su vez la interacción entre las proteínas de estas dos familias está mediada por los dominios QLQ, en los factores de transcripción GRF, y SNH, en los coactivadores GIF.
Dentro del proyecto estudiamos:
-La formación de los complejos mediados por los dominios QLQ y SNH. Ambos dominios son desordenados en su forma libre, y su interacción se produce a través de un mecanismo poco usual de plegamiento sinérgico mutuo.
-La estructura de los dominios WRC y su modo de reconocimiento de secuencias de ADN específicas.
-La presencia de elementos de reconocimiento molecular en las extensas regiones desordenadas de la proteína y su participación en la regulación de la conformación global de los complejos.
Optimización de enzimas
Las enzimas aplican en distintos procesos industriales modernos, donde se aprovecha su especificidad y su calidad en la catálisis de reacciones químicas. En general, los medios de reacción que se utilizan difieren sustancialmente respecto del medio celular o extracelular en el cual las enzimas han evolucionado naturalmente. El uso eficiente de enzimas en estos medios hostiles ha requerido en muchos casos de su modificación a partir de ingeniería de proteínas, llevando a variantes mejor adaptadas, que mantienen su funcionalidad.
Nuestros proyectos en esta línea buscan obtener variantes de enzimas con mayor estabilidad y actividad que las presentes en la naturaleza. Nuestro objeto de estudio son fosfolipasas C bacterianas. Estas enzimas se utilizan en el proceso industrial de desgomado enzimático de aceite, de gran relevancia económica en la región del Gran Rosario. Aplicamos métodos de combinación de secuencias conocidas y de aprendizaje automático para generar nuevas secuencias de proteínas. Evaluamos la estabilidad y la actividad de variantes seleccionadas mediante técnicas espectroscópicas y bioquímicas. A través de los resultados obtenidos, abordamos cuestiones aun no resueltas sobre los fundamentos de la estabilidad y reactividad de proteínas.
Integración de señales que controlan el crecimiento celular y desarrollo temprano en plantas
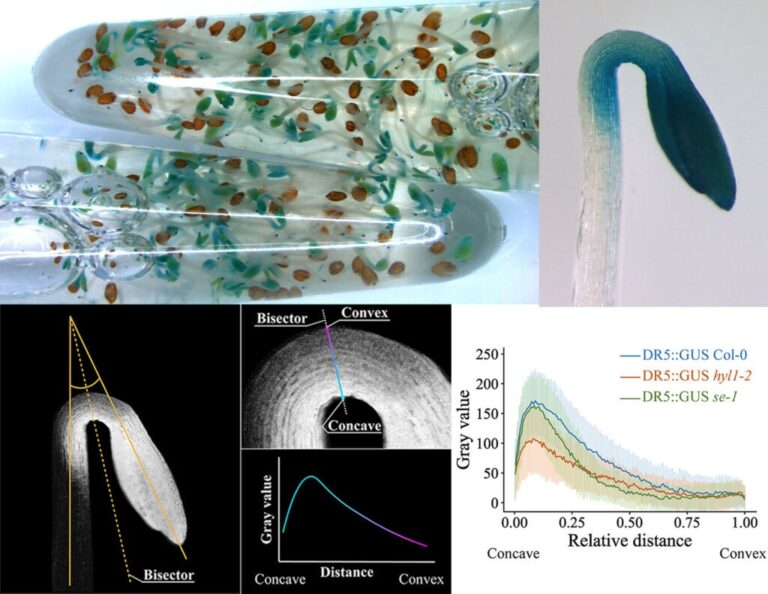
Las múltiples señales ambientales, junto a señales endógenas, son percibidas e integradas por las especies vegetales mediante sofisticados mecanismos moleculares para elaborar una respuesta idónea en el crecimiento y desarrollo. En el laboratorio utilizamos como modelo de estudio el desarrollo temprano de la planta modelo Arabidopsis thaliana desde que germina y crece en oscuridad, proceso denominado escotomorfogénesis, hasta que encuentra la luz para sintetizar y activar la maquinaria fotosintética para ser autosuficiente (fotomorfogénesis). Este proyecto comprende tres líneas de trabajo que abordan cómo las plantas integran señales externas y endógenas para decidir el crecimiento de sus órganos y activar sus programas de desarrollo:
1-Mecanismos moleculares que controlan el desarrollo temprano. Convergencia de señales de luz/oscuridad, biogénesis de microARNs y auxinas para el correcto desarrollo escotomorfogénico.
2-Conservación funcional y mecanística del regulador maestro HY5. Abordamos la conservación de la actividad del factor de transcripción HY5 entre Arabidopsis y el árbol modelo álamo, especie clave para la generación de biomasa ante el cambio climático.
3-Arquitectura celular en el control de los procesos de crecimiento vegetal. Estudiamos la interdependencia de los microtúbulos corticales, los cuales condicionan el crecimiento de las células vegetales, con las proteínas señalizadoras de luz/oscuridad y las auxinas.
IMÁGENES DE NUESTRAS INVESTIGACIONES
PUBLICACIONES Y PATENTES
Characterization of a novel thermostable phospholipase C from T. kodakarensis suitable for oil degumming.
Characterization of a novel thermostable phospholipase C from T. kodakarensis suitable for oil degumming.
HYPONASTIC LEAVES 1 is required for proper establishment of auxin gradient in apical hooks.
HYPONASTIC LEAVES 1 is required for proper establishment of auxin gradient in apical hooks.
Dual function of HYPONASTIC LEAVES 1 during early skotomorphogenic growth in Arabidopsis.
Dual function of HYPONASTIC LEAVES 1 during early skotomorphogenic growth in Arabidopsis.
Conformational sampling of the intrinsically disordered dsRBD-1 domain from Arabidopsis thaliana DCL1.
Conformational sampling of the intrinsically disordered dsRBD-1 domain from Arabidopsis thaliana DCL1.
The key role of electrostatic interactions in the induced folding in RNA recognition by DCL1-A.
The key role of electrostatic interactions in the induced folding in RNA recognition by DCL1-A.
Sede CCT Rosario
Ocampo y Esmeralda, Predio CONICET-Rosario
2000 Rosario, Santa Fe, Argentina
Tel. 54-341-4237070 / 4237500 / 4237200
Sede Facultad de Ciencias Bioquímicas y Farmacéuticas
Universidad Nacional de Rosario - Suipacha 531
2000 Rosario, Santa Fe, Argentina
Tel. +54 341 4350596 / 4350661 / 4351235
🔬 El IBR suma 9 proyectos seleccionados en Investigación Orientada 2025 de @ProduccionSF y @CienciaSantaFe.
Biotecnología, salud y sostenibilidad para fortalecer el vínculo entre ciencia, innovación y desarrollo territorial.

