GRUPOS DE INVESTIGACIÓN
Mecanismos de Resistencia Bacteriana a Antimicrobianos
RESUMEN

Sede:
Email:
- Cavatorta, Matias
LÍNEA DE INVESTIGACIÓN
La multirresistencia en Acinetobacter baumannii desde una óptica multifactorial. Evolución y diseminación de plásmidos portando genes de carbapenemasas tipo-OXA en cepas clínicas locales de este patógeno. Biogénesis de estas enzimas
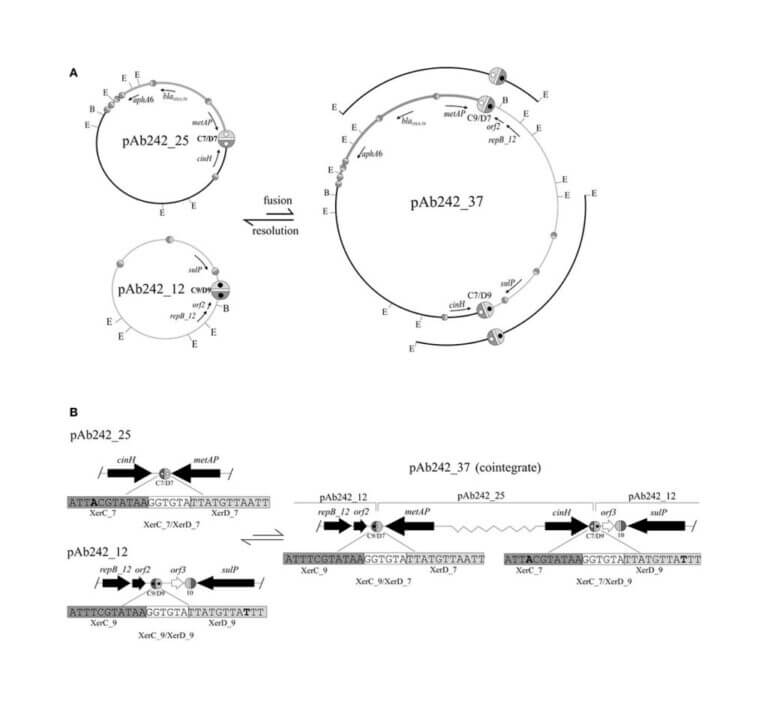
La resistencia antimicrobiana en A. baumannii es multifactorial, e incluye la producción de oxacilinasas (OXA) con actividad carbapenemasa adquiridas mediante transferencia horizontal de genes (THG). El objetivo es dilucidar las bases moleculares de la evolución y diseminación de genes blaOXA en clones multirresistentes de A. baumannii. El gen blaOXA-58, principal responsable de resistencia a carbapenemes en cepas locales, se encuentra bordeado por sitios de reconocimiento de recombinasas específicas de sitio XerC/D. Postulamos que dichos sitios posibilitarían la movilización de genes blaOXA a otras regiones genómicas o plásmidos mediante eventos de recombinación. Estudiamos así el rol de los sitios XerC/D plasmídicos en la evolución de plásmidos de resistencia, así como mecanismos de THG en su diseminación en la población clínica de A. baumannii y otras especies del género. OXA-58 es asimismo exportada al periplasma como una lipoproteína, permitiendo su secreción como “cargo” en vesículas de membrana externa, contribuyendo a la multirresistencia bacteriana. La biogénesis de dichas vesículas, así como su rol en la misma, son materia de estudio. Investigadores responsables: J. Morán Barrio – A. Limansky – A. Viale.
Pseudomonas (grupo P. putida) y Acinetobacter bereziniae como reservorios ambientales de genes de metalo-beta-lactamasas blaVIM-2 y blaNDM-1 confiriendo resistencia a carbapenemes. Plataformas genéticas responsables de su diseminación e impacto clínico.
La eclosión de cepas nosocomiales resistentes a carbapenemes se debe esencialmente a eventos intragenómicos que involucran la movilización de genes codificantes para distintas carbapenemasas incluyendo diversas metalo-beta-lactamasas (MBL), sumado a la THG. Así, resulta relevante dilucidar la contribución de cada uno de estos eventos a la evolución de la misma. En este contexto, y bajo la hipótesis de que los genes de resistencia son seleccionados en bacterias sometidas a presión antimicrobiana, es esencial caracterizar plataformas genéticas que portan genes de MBL tanto de amplia diseminación (blaVIM-2), como recientes (blaNDM-1). Estudiamos, con este objetivo, las bases moleculares de la diseminación de genes codificantes de estas enzimas en microorganismos que constituyen reservorios nosocomiales, con énfasis en especies de los géneros Pseudomonas y Acinetobacter, como Pseudomonas grupo putida y A. bereziniae. Conocer la evolución y diseminación de estos genes desde reservorios bacterianos a patógenos u oportunistas multirresistentes contribuirá al control de infecciones nosocomiales. Investigadores responsables: A. Limansky – A. Viale – J. Morán Barrio.
Contribución de proteínas secretadas en vesículas de membrana externa a la fisiopatología de Acinetobacter baumannii
La patogénesis de A. baumannii está asociada a la condición de multirresistencia (MR) y a factores de virulencia poco caracterizados. Existe una creciente evidencia de que las vesículas de membrana externa (VMEs) son vehículos para transportar proteínas bacterianas que contribuyen al establecimiento de la infección. Nuestro objetivo es estudiar la contribución de proteínas de secreción, con especial énfasis en lipoproteínas, a la sobrevida de este patógeno oportunista a distintas condiciones de estrés que mimeticen el contacto con el hospedador. Nos centramos en proteínas secretadas en asociación a VMEs en A. baumannii, con el fin de aumentar significativamente nuestra comprensión de su rol en su fisiopatología. El abordaje de los mecanismos de virulencia subyacentes en este importante patógeno nosocomial permitirá revelar nuevos blancos para el desarrollo de drogas terapéuticas. Investigador responsable: J. Morán Barrio.
IMÁGENES DE NUESTRAS INVESTIGACIONES
PUBLICACIONES Y PATENTES
Pseudomonas putida group species as reservoirs of mobilizable Tn402-like class 1 integrons carrying blaVIM-2 metallo-β-lactamase genes.
Pseudomonas putida group species as reservoirs of mobilizable Tn402-like class 1 integrons carrying blaVIM-2 metallo-β-lactamase genes.
What do we know about plasmids carried by members of the Acinetobacter genus?
What do we know about plasmids carried by members of the Acinetobacter genus?
Microevolution in the major outer membrane protein OmpA of Acinetobacter baumannii.
Microevolution in the major outer membrane protein OmpA of Acinetobacter baumannii.
Site-specific recombination at XerC/D sites mediates the formation and resolution of plasmid co-integrates carrying a blaOXA-58- and TnaphA6-resistance module in Acinetobacter baumannii.
Site-specific recombination at XerC/D sites mediates the formation and resolution of plasmid co-integrates carrying a blaOXA-58- and TnaphA6-resistance module in Acinetobacter baumannii.
Characterization of the diverse plasmid pool harbored by the blaNDM-1-containing Acinetobacter bereziniae HPC229 clinical strain.
Characterization of the diverse plasmid pool harbored by the blaNDM-1-containing Acinetobacter bereziniae HPC229 clinical strain.
Sede CCT Rosario
Ocampo y Esmeralda, Predio CONICET-Rosario
2000 Rosario, Santa Fe, Argentina
Tel. 54-341-4237070 / 4237500 / 4237200
Sede Facultad de Ciencias Bioquímicas y Farmacéuticas
Universidad Nacional de Rosario - Suipacha 531
2000 Rosario, Santa Fe, Argentina
Tel. +54 341 4350596 / 4350661 / 4351235
Dirigido por las Dras. Julia Cricco y Pamela Cribb, el equipo reúne expert@s en biología, química, veterinaria y comunicación. La propuesta combina ciencia rigurosa y compromiso social.
#UnaSolaSalud #InvestigaciónAplicada

